Книга: Приспособиться и выжить!
Назад: Рассматриваем радугу
Дальше: Запутанная история возникновения цветового зрения
От молекул к деревьям
С тех пор как Дарвин предположил, что эволюционное развитие организмов можно представить в виде ветвящегося дерева, биологи все время рисуют подобные деревья для изображения родственных связей между ныне живущими и когда-то жившими организмами. Раньше подобные деревья строились на основании внешних признаков современных организмов или ископаемых остатков древних организмов. Однако довольно часто выяснялось, что внешнее сходство или его отсутствие уводит не в ту сторону или по меньшей мере вызывает споры между биологами, поэтому практически для всех основных групп организмов было создано несколько генеалогических деревьев.
Несколько десятилетий назад для определения степени родства между видами ученые начали использовать последовательности биологических молекул. Гены наследуются, и это означает, что последовательности генов и кодируемых ими белков передаются от родителей детям. Происходящие в этих последовательностях изменения также передаются потомкам на всем долгом пути эволюции. Таким образом, степень сходства ДНК может служить показателем близости родства между видами.
Теоретически для построения генеалогического древа биологи имеют в своем арсенале тысячи последовательностей ДНК или белков. На практике для выбора генов для построения древа используют несколько критериев отбора (в частности, стоимость анализа), так что обычно из многих генов-кандидатов выбирают лишь один или несколько. На основании выбранных последовательностей генов с помощью сложного математического и статистического аппарата строят генеалогическое древо. Это древо должно как можно точнее соответствовать имеющимся экспериментальным данным. Так как последовательности генов состоят всего из четырех знаков (A, C, G и T), при недостаточном количестве данных родство между организмами может быть определено неточно. Не следует думать, что с помощью любого гена можно получить один и тот же правильный или однозначный ответ. Поэтому один из способов проверить, насколько верно выстроено древо, — попытаться воспроизвести его на основании другой последовательности или более широкого круга последовательностей.
К счастью, был разработан совершенно новый метод определения родственных связей между видами. Он также основан на анализе ДНК, но учитывает не степень сходства последовательностей в целом, а наличие или отсутствие определенных маркеров в специфических участках ДНК. Эти маркеры возникают в результате случайного встраивания мусорной ДНК рядом с генами. Достаточно часто в ДНК встречаются специфические последовательности, называемые длинными диспергированными повторами (LINE) и короткими диспергированными повторами (SINE). Если такие повторы встраиваются в последовательность, они в ней остаются, поскольку не существует механизма их активного удаления. Последовательность этих элементов в составе ДНК является характерной для данного вида и передается по наследству всем потомкам. Это замечательный инструмент для построения генеалогического древа. Случаи встраивания подобных последовательностей встречаются очень редко, поэтому их присутствие в одном и том же участке ДНК двух разных видов может объясняться исключительно происхождением этих видов от общего предка. На том же принципе наследования маркеров в ДНК основано определение отцовства у людей. Анализ распределения нескольких маркерных элементов ДНК, возникших в разное время у разных организмов, дает биологам достаточно данных, чтобы абсолютно точно устанавливать родственные отношения между видами.
Мы вкратце ознакомились с сутью процесса построения генеалогического древа, но давайте не будем ставить точку. Мне хочется, чтобы, читая эту книгу, вы в полной мере оценили природу и качество имеющихся доказательств эволюции. Чтобы поверить и понять, нужно увидеть. Поэтому давайте посмотрим, как с помощью повторов SINE было построено такое важное генеалогическое древо, как древо человека и других приматов, а затем при помощи этого древа раскроем тайну происхождения цветового зрения.
Прежде всего при построении древа нужно обнаружить набор повторов SINE. Последовательность генома человека известна, что облегчает задачу: мы можем выбирать из тысяч коротких диспергированных повторов. В соответствующих участках ДНК других приматов ищут те же самые повторы.
Большинство SINE состоят примерно из 300 пар оснований, поэтому, если в ДНК какого-то организма содержится такой же SINE, как в ДНК человека, этот фрагмент ДНК будет на 300 пар оснований длиннее, чем в случае отсутствия SINE. На рис. 4.3 представлены данные, полученные Абдель-Халимом Салемом и его коллегами из университетов Луизианы и Юты.
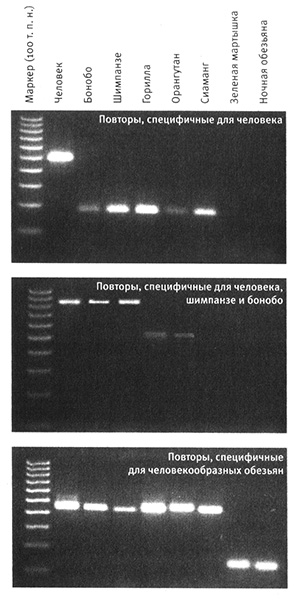
Рис. 4.3. Типирование ДНК и эволюция гоминидов. Наличие определенных повторов SINE в ДНК разных видов организмов определяют по яркой полосе на соответствующей дорожке в геле. Наличие одинаковых повторов у представителей разных видов указывает на их эволюционное родство. Из статьи Salem et al., 2003, Proceedings of the National Academy of Science 100:12 787. © 2003 by the National Academy of Sciences, USA.
Наличие или отсутствие SINE определяется по положению полос ДНК в геле, используемом для разделения фрагментов ДНК разного размера. Один из повторов, представленных на рисунке, специфичен только для человека, второй является общим для человека, карликовых шимпанзе (бонобо) и шимпанзе, а третий — для человека, бонобо, шимпанзе, горилл, орангутанов и сиамангов (сростнопалых гиббонов). Среди 100 с лишним повторов были выявлены такие, которые являются общими для разных групп приматов, в том числе для всех шести исследованных видов человекообразных обезьян, а также лишь для пяти, четырех и т. д. видов человекообразных. Также были выявлены повторы, которые являются общими лишь для бонобо и шимпанзе, и уникальные для человека, бонобо или шимпанзе. Ни один из исследованных повторов не был обнаружен в геноме ночных (совиных) обезьян. Количество повторов, являющихся общими для разных видов, отражает степень родства этих видов, и может служить для построения генеалогического древа (рис. 4.4).
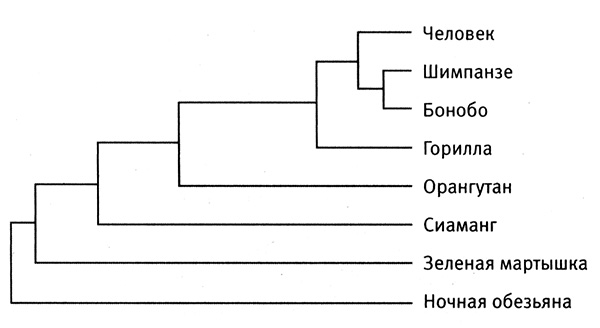
Рис. 4.4. Эволюционное древо гоминоидов. Эволюционное родство между гоминидами и обезьянами, установленное на основании анализа ДНК. На основании данных из Salem et al., 2003, Proceedings of the National Academy of Science 100:12 787.
Из этого древа следует, что ближайшими родственниками человека являются шимпанзе, ближайшими родственниками шимпанзе — бонобо, а ветви горилл и орангутанов отделились от древа до появления последнего общего предка человека и шимпанзе. Достоверность этой информации не подлежит сомнению.
Теперь давайте с помощью этого древа займемся изучением происхождения цветового зрения и опсинов.

